BioPathBot
Aller à la navigation
Aller à la recherche
Description
Depuis une databiographie suivant le format utilisé sur le wiki (ex: 1890.03.19 / Genève), le BioPathBot est capable d'extraire les informations temporelles et spatiales. Ces deux données forment un tuple et sont parsées de façon suivante :
- La date est formée d'une année, d'un mois et d'un jour. Si des informations venaient à manquer, la date est complétée par défaut avec le mois de janvier et le premier du mois.
- Le lieu est transformé en coordonnées géographiques (utilisant la librairie python geopy)
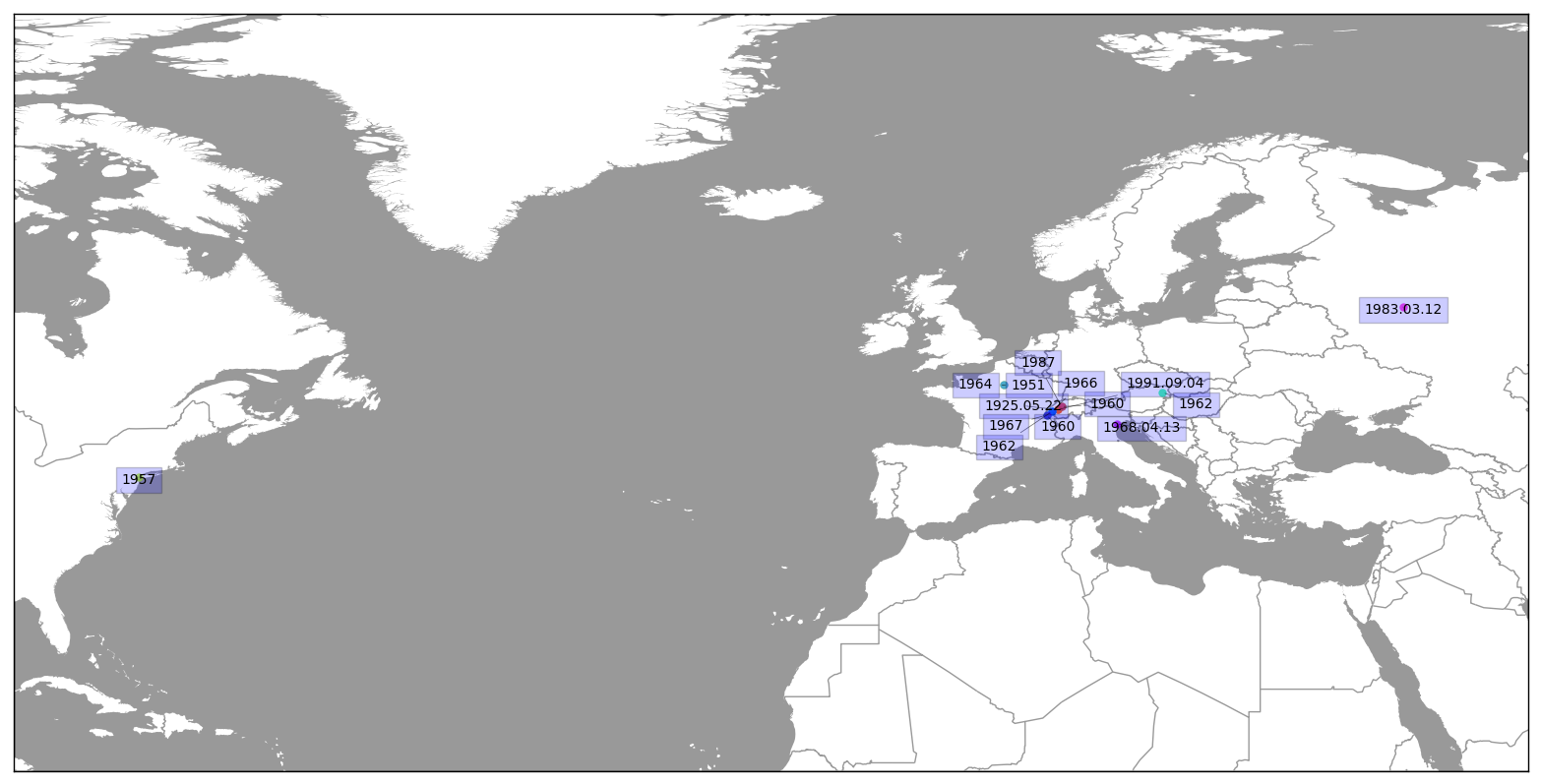
Ainsi transformées et triées chronologiquement, ces données permettent de générer une carte utilisant basemap, où sont dessinés des points pour marquer les lieux, ainsi que des traits mettant en avant la trajectoire de la personne. Cette carte est ensuite importée sur wikipast puis insérée sur une page annexe.
Par exemple la carte de Jean Tinguely se visualise comme:
Critique
- Peu importe le type de page (une page concernant une personne, un hypermot...), une carte est générée. Donc si une page ne relate pas la vie d'une personne, la carte aura peu de sens.
- Avec l'algorithme actuel, les dates et les lieux sont parfaitement extraits et placé sur la carte. Cependant, si les données ne sont pas suffisamment précises, elles seront approximées (1980 devient 1980.01.01 tandis que le point géographique correspondant à la Suisse sera son centre géographique). Cela peut mener à des erreurs de trajectoire lors du tri chronologique ou simplement à une position différente de la réalité sur la carte.
- S'il existe des événements post-mortem mais que la mort de la personne n'est pas mentionnée ou ne suit pas la convention de l'hypermot Décès, ces données seront aussi extraites et placées sur la carte, alors même que la personne ne se déplace pas.
- Si des événements sont spatialement trop proches, ils vont se superposer sur la carte et il deviendra difficile de retracer les événements d'une façon visuellement claire. Par exemple, pour un trait entre Berne et Lausanne, il sera difficile de juger si une personne a fait plusieurs fois ce même déplacement ou ne l'a fait qu'une seule fois au cours de sa vie.
Code
import urllib.request import requests from bs4 import BeautifulSoup import re import math import datetime from geopy.geocoders import Nominatim from mpl_toolkits.basemap import Basemap import matplotlib.pyplot as plt from colorsys import hsv_to_rgb from matplotlib.colors import rgb2hex SEGMENTS = 100 # draw plots inline rather than in a seperate window %matplotlib inline # draw plots bigger plt.rcParams["figure.figsize"] = [20.0, 10.0] user='BioPathBot' passw='chkiroju' baseurl='http://wikipast.epfl.ch/wikipast/' summary='Wikipastbot update' names=['Jean Tinguely'] # Login request payload={'action':'query','format':'json','utf8':,'meta':'tokens','type':'login'} r1=requests.post(baseurl + 'api.php', data=payload) #login confirm login_token=r1.json()['query']['tokens']['logintoken'] payload={'action':'login','format':'json','utf8':,'lgname':user,'lgpassword':passw,'lgtoken':login_token} r2=requests.post(baseurl + 'api.php', data=payload, cookies=r1.cookies) #get edit token2 params3='?format=json&action=query&meta=tokens&continue=' r3=requests.get(baseurl + 'api.php' + params3, cookies=r2.cookies) edit_token=r3.json()['query']['tokens']['csrftoken'] edit_cookie=r2.cookies.copy() edit_cookie.update(r3.cookies) #setup geolocator geolocator = Nominatim(timeout=10) # upload config def uploadMap(filename): # read local file upload_file = open(filename,"rb") upload_contents = upload_file.read() upload_file.close() # setting parameters for upload # ref: https://www.mediawiki.org/wiki/API:Upload payload={'action':'upload','filename':filename, 'ignorewarnings':1, 'token':edit_token} files={'file':upload_contents} # upload the image print("Uploading file to %s via API..." % (baseurl+"index.php/Fichier:"+filename)) r4=requests.post(baseurl+'api.php',data=payload,files=files,cookies=edit_cookie) # in case of error print the response #print(r4.text) def addToPage(name, img): title = name + " BioPathBot" content = "Fichier:"+ img +"" pageToChange = requests.post(baseurl+'api.php?action=query&titles='+title+'&export&exportnowrap') payload={'action':'edit','assert':'user','format':'json','utf8':,'text':content,'summary':summary,'title':title,'token':edit_token} r4=requests.post(baseurl+'api.php',data=payload,cookies=edit_cookie) # @TODO not get post-mortem data -> check Décès # BioPathBot : add line of databiographie to the right page (time and space) def getDataFromPage(name): data = [] result=requests.post(baseurl+'api.php?action=query&titles='+name+'&export&exportnowrap') soup=BeautifulSoup(result.text, "lxml") #soup=BeautifulSoup(result.text) code= for primitive in soup.findAll("text"): code+=primitive.string # split on list (*) lines = code.split("*") for line in lines : # add breaking lines (otherwise will be appened directly in one line) line = "\n\n"+line # get date if exist date = re.findall("((?<=\[\[)\d*(\.*\d*\.*\d*)*(?=\]\]))",line) dateToAdd = "" if len(date) != 0 : dateToAdd = date[0][0] # get place if exist place = re.findall("(?<=\/\s\[\[)[A-zÀ-ÿ]*(?=\]\])",line) location = "" if len(place) != 0: placeToAdd = place[0] location = geolocator.geocode(placeToAdd) # if both the date and the location are available, append in data array if dateToAdd and location: dataToAdd = [location.longitude,location.latitude]; data.append(dataToAdd); return data # finds the minimal and maximal longitude and latitude def findCorners(pts): minlon = maxlon = pts[0][0] minlat = maxlat = pts[0][1] for p in pts: currlon = p[0] if currlon<minlon: minlon = currlon elif currlon>maxlon: maxlon = currlon currlat = p[1] if currlat<minlat: minlat = currlat elif currlat>maxlat: maxlat = currlat return [minlon, maxlon, minlat, maxlat] # draws the map, some points and the lines def drawmap(pts, filename, export=False): n_pts = len(pts) corners = findCorners(pts) m = Basemap(llcrnrlon=corners[0]-1, llcrnrlat=corners[2]-1, urcrnrlon=corners[1]+1, urcrnrlat=corners[3]+1, resolution='i') m.drawmapboundary(fill_color='0.6') m.drawcountries(linewidth=1.0, color='0.6') m.fillcontinents(color='white', lake_color='white') for i in range(n_pts-1): # draw lines for j in range(SEGMENTS): start = pts[i] + (pts[i+1]-pts[i])*(j/SEGMENTS) end = pts[i] + (pts[i+1]-pts[i])*((j+1)/SEGMENTS) m.plot([start[0], end[0]], [start[1], end[1]], color=hsv_to_rgb((i+j/SEGMENTS)/n_pts, 1, 1)) for i in range(n_pts): # draw points m.plot(pts[i][0], pts[i][1], marker='o', color=hsv_to_rgb(i/n_pts, 1, 1), fillstyle='full', markeredgewidth=0.0) if export: plt.savefig(filename, bbox_inches='tight') plt.show() for name in names: image_filename = (name + "_biopath.png").replace(" ","_") data = getDataFromPage(name) drawmap(np.array(data), image_filename, True) uploadMap(image_filename) addToPage(name, image_filename)
Groupe
| Nom et Prénom | Pseudo |
|---|---|
| Christophe Badoux | Christophe |
| Julien Burkhard | JulienB |
| Kim Lan Phan Hoang | Kl |
| Robin Lang | Roblan11 |